Einzelnukleotid-Polymorphismus
Ein Einzelnukleotid-Polymorphismus (SNP, ausgeprägter Schnipsel; Pluralschnipsel) ist eine DNA-Sequenzvariation in einer Population. Ein SNP ist nur ein einziger Nukleotidunterschied im Genom.
Zum Beispiel sind sequenzierte DNA-Fragmente von zwei Personen, AAGCCTA bis AAGCTTA, in einem einzigen Nukleotid unterschiedlich. In diesem Fall sagen wir, dass es zwei Allele gibt. Fast alle gängigen SNPs haben nur zwei Allele. SNPs kommen am häufigsten in Regionen der DNA vor, die das Überleben des Organismus nicht beeinflussen: andernfalls würden sie durch natürliche Selektion aussortiert werden. Auch andere Faktoren, wie die genetische Rekombinations- und Mutationsrate, können die SNP-Dichte beeinflussen. Es gibt Unterschiede zwischen menschlichen Populationen, so dass ein SNP-Allel, das in einer geografischen oder ethnischen Gruppe häufig vorkommt, in einer anderen viel seltener sein kann.
Diese genetischen Variationen zwischen Individuen (insbesondere in nicht-kodierenden Teilen des Genoms) werden manchmal bei der Erstellung von DNA-Fingerabdrücken ausgenutzt, die in der forensischen Wissenschaft verwendet werden. Diese genetischen Variationen verursachen auch Unterschiede in unserer Anfälligkeit für Krankheiten. Die Schwere der Krankheit und die Art und Weise, wie unser Körper auf Behandlungen reagiert, sind ebenfalls Manifestationen genetischer Variationen. So ist zum Beispiel eine einzige Basenmutation im APOE (Apolipoprotein E)-Gen mit einem höheren Risiko für die Alzheimer-Krankheit verbunden.
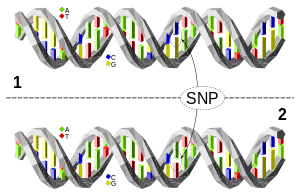
DNA-Molekül 1 unterscheidet sich von DNA-Molekül 2 an einer einzigen Basenpaar-Stelle (ein C/A-Polymorphismus)
Fragen und Antworten
F: Was ist ein SNP?
A: Ein Einzelnukleotid-Polymorphismus ist eine DNA-Sequenzvariation in einer Population, die durch einen einzigen Nukleotidunterschied im Genom gekennzeichnet ist.
F: Was bedeutet SNP?
A: SNP steht für Single Nucleotide Polymorphism (Einzelnukleotid-Polymorphismus).
F: Wie viele Allele haben häufige SNPs?
A: Die meisten häufigen SNPs haben nur zwei Allele.
F: Wo kommen SNPs am häufigsten in der DNA vor?
A: SNPs treten am häufigsten in Bereichen der DNA auf, die keinen Einfluss auf das Überleben des Organismus haben.
F: Welche Faktoren können die SNP-Dichte beeinflussen?
A: Faktoren wie die genetische Rekombination und die Mutationsrate können die SNP-Dichte ebenfalls beeinflussen.
F: Welche praktischen Anwendungen gibt es für genetische Variationen zwischen Individuen?
A: Genetische Variationen zwischen Individuen (insbesondere in nicht kodierenden Teilen des Genoms) werden manchmal für den DNA-Fingerabdruck genutzt, der in der Forensik verwendet wird. Diese genetischen Variationen verursachen auch Unterschiede in unserer Anfälligkeit für Krankheiten.
F: Was ist ein Beispiel für eine genetische Variation, die mit einem höheren Risiko für eine bestimmte Krankheit verbunden ist?
A: Eine einzelne Basenmutation im APOE (Apolipoprotein E)-Gen wird zum Beispiel mit einem höheren Risiko für die Alzheimer-Krankheit in Verbindung gebracht.
Suche in der Enzyklopädie